آموزش طراحی پرایمر qPCR

طراحی پرایمر qPCR یک مرحله حیاتی در بررسی بیان ژن و تجزیه تحلیلهای آن میباشد. یک طراحی پرایمر مناسب در فرآیند بررسی بیان ژن میتواند کارآیی pcr را چندین برابر بالا ببرد. همچنین، در مواردی که از روش One step RT-PCR استفاده میشود استفاده از توالی پرایمر ناکارآمد میتواند روند تولید cDNA را با اختلال مواجه کند.
با توجه به موارد فوق، ارزش صرف زمان برای طراحی پرایمر qPCR قابل توجیه میباشد. برای درک اینکه یک پرایمر خوب چیست مهم است که به یاد داشته باشید که SYBR® Green چگونه کار میکند. رنگ سایبر گرین به هر dsDNA موجود در مخلوط واکنش متصل میشود، بنابراین محصولات غیر اختصاصی همزمان با نمونه تکثیر خواهد شد که داده های نامعتبری به همراه خواهد داشت.
عوامل دیگری که باید در نظر گرفت تشکیل پرایمر دایمرها و کارایی پرایمر است. راندمان (میزان عملکرد پرایمرها) واکنش qPCR، باید ۹۰ تا ۱۰۰ درصد باشد. پرایمرها با کارایی بالا حساسیت کمّی سازی را افزایش می دهند. عواملی که بر کارایی qPCR تاثیر میگذارند شامل طول امپلیکون و کیفیت پرایمر است. به طور خلاصه، کلید طراحی پرایمرهای خوب مبتنی بر SYBR® Green، یافتن یک جفت پرایمری است که برای ژن مورد نظر به صورت اختصاصی بوده و دایمر پرایمر تولید نکند، دانستن پارامترهای رایج، که در اکثر نرم افزارهای طراحی پرایمر قابل تنظیم هستند، میتواند به دستیابی به این امر کمک کند.
مطالب پیشنهادی: انواع واکنش زنجیره ای پلیمراز PCR – عیب یابی Real time pcr
ابزار NCBI Primer-BLAST به طور گسترده برای طراحی پرایمر در qPCR استفاده می شود. بسیاری دیگر از ابزارهای طراحی پرایمر به صورت آنلاین و آفلاین در دسترس هستند، از جمله آن ها می توان به primer3، UCSC, Oligo7,Oligo Analyzer اشاره کرد.
در ادامه مراحل اصلی آموزش طراحی پرایمر qPCR با استفاده از ابزار قابل دسترس برای همه کاربران به نام NCBI Primer-BLAST به طور مختصر و کارآمد آورده شده است، در برنامههای طراحی پرایمر دیگر، مراحل مشابه خواهند بود. به طور کلی این اطلاعات ایدهای از پارامترهایی که باید حین طراحی رعایت کرد را به کاربر ارائه میدهد.
طراحی پرایمر qPCR با ابزار Primer-BLAST
Primer-BLAST پلت فرم طراحی پرایمر آنلاین رایگان NCBI است که پرایمرهای PCR را با استفاده از برنامه Primer3 طراحی می کند و همزمان با آن احتمال اتصال پرایمرها به مناطق نامشخص ژنوم ارگانیسم مورد نظر را از طریق الگوریتم BLAST بررسی میکند.
مراحل طراحی پرایمر با استفاده از Primer Blast
ژن خود را جستجو کنید
مهمترین مرحله از آموزش طراحی پرایمر qPCR جستجوی توالی نوکلئوتیدی ژن مورد نظر در NCBI است. برای این کار به پایگاه داده NCBI رفته گزینه جستجو را بروی Nucleotide قرار داده و ژن مورد نظر را جستجو می کنیم. از آنجایی که پایگاه داده NCBI بسیار گسترده است، باید جستجوی خود را به بهترین شکل ممکن محدود کنیم. برای این منظور ، اسم کامل ژن خود را همراه با گونه مورد نظر را در کادر جستجو وارد می کنیم. به عنوان مثال، ژن اینترلوکین-۶ را جستجو می کنیم. در صفحه نتایج نیز میتوانید از نوار کناری سمت چپ صفحه برای فیلتر کردن جستجوی خود استفاده کنید. برای مشخص کردن گونه مورد نظر در زیر عنوان Organism مورد Homo sapiens را انتخاب (اگر این گزینه موجود نبود با کلیک بروی Customize و جستجو گزینه مورد نظر را انتخاب و اضافه می کنیم) برای بررسی بیان ژن در زیر عنوان Molecular types مورد mRNA را انتخاب می نمائیم. البته، همه ی این موارد می توان متناسب با ژن مورد نظر شما تغییر کند.
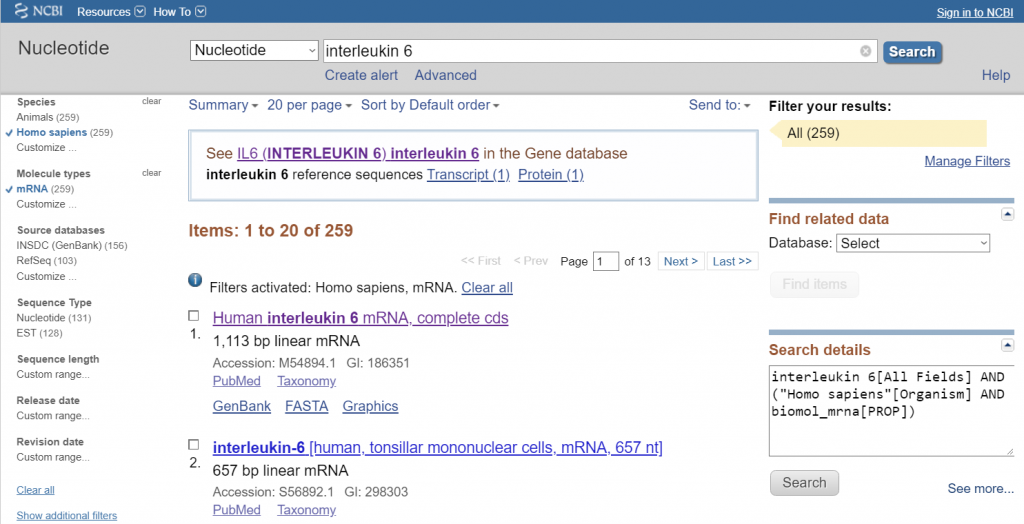
انتخاب ژن مورد نظر و واریانت های آن در Primer-BLAST
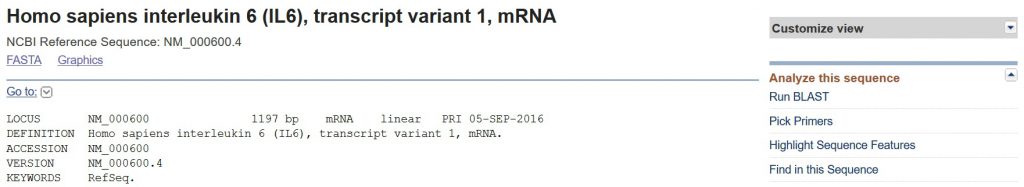
در این قسمت چیزی که می خواهید به دنبال آن باشید، توالی mRNA ژن مورد نظر شما است. توالی معمولا دارای یک شماره دسترسی است که با “_NM” شروع می شود. توجه داشته باشید که برخی از ژن ها رونوشتهای متعددی نیز دارند، بنابراین مطمئن شوید که توالی mRNA صحیح را برای ژن و رونوشت مورد نظر خود انتخاب کرده اید. برخی از پرایمرها نیز دارای “cd” هستند به این معنی که توالی مربوط به DNA است که به محصول پروتئینی ترجمه می شود پس در طراحی دقت کنید که cd ها را انتخاب نکنید، زیرا ما به اطلاعاتی در مورد اینترون ها نیاز داریم. هنگامی که توالی مورد نظر خود را پیدا کردید، روی آن کلیک کنید. برای مثال: «Homo sapiens interleukin 6 (IL6), transcript variant 1, mRNA».
Primer-BLAST را باز کنید
در صفحه توالی، اطلاعات زیادی در مورد توالی ژن مورد نظر، از جمله توالی خام مربوط به آن را خواهید دید. کاری که می خواهیم انجام دهیم این است که اکنون Primer-BLAST را برای طراحی پرایمر PCR برای ژن مورد نظر آماده کنیم. برای انجام این کار، در نوار ابزار کناری سمت راست در زیر تب “Analyze this sequence” گزینه ی “Pick Primers” را انتخاب کنید.
معیارهای پرایمرهای مورد نظر را تعیین کنید
در این بخش معیارهای کلیدی در طراحی پرایمر قابل تنظیم است. این معیارهای کلیدی در مطلب اصول طراحی پرایمر به طور کامل شرح داده شده است. چندین بخش در صفحه Primer-BLAST وجود دارد، به دلیل اهمیت این قسمت هر کدام از موارد در ادامه به صورت جداگانه توضیح داده می شود.
مطالب پیشنهادی: اصول طراحی پرایمر – طراحی پرایمر miRNA
تعییین الگوی PCR در طراحی پرایمر qPCR
بخش اول شامل شماره دسترسی است. اگر مراحل قبل را به درستی انجام داده باشید، شماره دسترسی پرایمر برای شما در قسمت Enter accession, gi, or FASTA sequence وارد می شود. برای مثال ما، شماره دسترسی برای ژن مورد نظر «NM_000600.4» است. این یک کد منحصر به فرد است که به Primer-BLAST میگوید پرایمرهایی را بر اساس توالی «Homo sapiens interleukin 6 (IL6)، نسخه رونوشت نوع ۱، mRNA» طراحی کند. در صورت تمایل می توانید یک توالی را وارد یا یک توالی با فرمت FASTA آپلود کنید. همچنین، می توانید طیف وسیعی را برای طراحی مجموعه پرایمر مشخص کنید. اما ما این کار را انجام نمی دهیم.
تعیین پارامترهای پرایمر
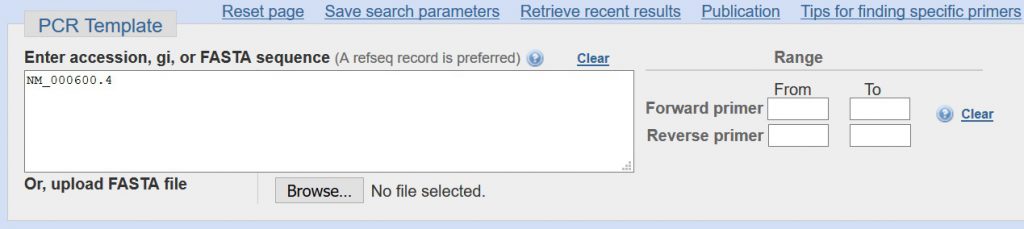
در بخش Primer Parameters، می توانید توالی پرایمرهای خود را وارد کنید. این دو مورد برای زمانی است که پرایمر را طراحی کرده اید و به دنبال اطلاعات مربوط به نحوه اتصال و ویژگی های انحصاری پرایمر خود هستید. ولی در مواردی که توالی ژن مورد نظر را نمی دانید قسمت مربوط به توالی پرایمر مورد نظر را خالی بگذارید.
در ادامه طول مورد نظر را برای توالی تکثیر شونده تعیین کنید. درReal-time PCR، اندازه مطلوب محصول PCR بین ۷۰ تا ۲۰۰ جفت باز است. بنابراین پارامتر PCR product size قسمت Max را به “۲۰۰” تغییر دهید. البته این اعداد مطلق نیست و میتوان بسته به نوع کار، آن را کم یا زیاد نمود. در این حالت ما شرایط بهینه یک واکنش را در نظر می گیریم.
گزینه of primers to return# به سادگی تعداد جفت پرایمرهایی است که میخواهید Primer-BLAST بر اساس معیارهای شما ایجاد کند را مشخص کنید. به صورت نرمال بهتر است این گزینه در ۱۰ توالی تنظیم شود. در پایین قسمت Primer Parameters می توانید محدوده دمای ذوب پرایمر (Tm) را مشخص کنید. تنظیمات پیش فرض برای این کار به ترتیب حداقل و حداکثر Tm 57 C° و ۶۳ C° با دمای بهینه ۶۰ C° تعریف شده است. “Max Tm difference” تفاوت در Tm بین پرایمرهای فوروارد و ریورس را نشان میدهد که در این جا ۳ درجه سانتی گراد تنظیم میشود.
برای real-time PCR، از دمای annealing ی ااتصال حدود ۶۰ درجه سانتیگراد برای واکنش PCR خود استفاده می کنیم. بنابراین، می خواهیم دمای ذوب بهینه پرایمر را به ۶۲ درجه سانتیگراد افزایش می دهیم. به عنوان یک قانون کلی، دمای annealing واکنش PCR تقریباً ۳ تا ۵ درجه سانتیگراد کمتر از Tm جفت پرایمر شما باید باشد. البته این عدد یک تقریب است. بنابراین، یک جفت پرایمر با Tm حدود ۶۲ درجه سانتیگراد، از نظر تئوری باید دمای annealing 57 – 59 درجه سانتیگراد داشته باشد.
انتخاب اگزون/اینترون در طراحی پرایمر qPCR
انتخاب نوع اگزون/اینترون در روند طراحی و اختصاصیت توالی همیشه یک نگرانی عمده برای کاربران بوده است. به همین منظور برای افزایش اختصاصیت راه کارهای متعددی پیشنهاد میشود.
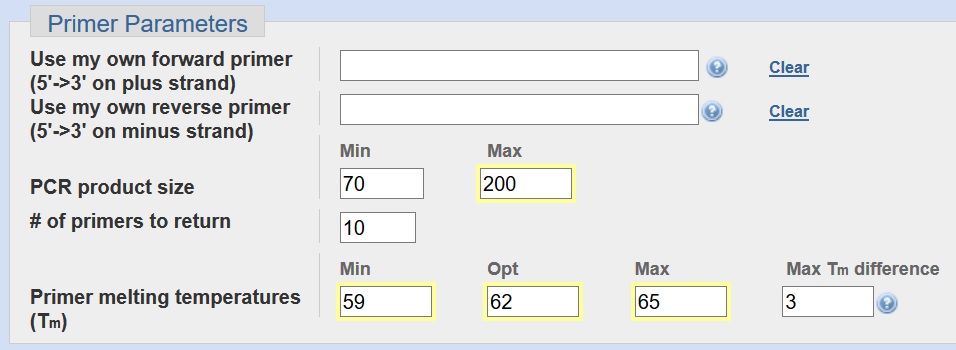
برای طراحی پرایمر qPCR با ویژگی «exon junction span»، میتوانید از Primer-BLAST بخواهید یک پرایمر برای اتصال به دو اگزون طراحی کند. این بدان معنی است که بخشی از پرایمر به یک اگزون متصل میشود، در حالی که بقیه به اگزون مجاور متصل می شود. میتوانید تعیین کنید که چه تعداد نوکلئوتید پرایمر به هر اگزون در بخش « exon junction match» متصل شود. با طراحی پرایمر با این روش (یک پرایمر در یک اگزون و پرایمر دیگر در اگزون دیگر) می توان توالی پرایمر را برای نمونه mRNA اختصاصی نمود و اگر نمونه در این حالت حاوی DNA باشد، به صورت اختصاصی، توالی ما از mRNA تکثیر خواهد شد.
با انتخاب گزینه ” intron inclusion” می توانید هر گونه آلودگی DNA ژنومی را در واکنش PCR خود شناسایی کنید، زیرا DNA ژنومی شامل اینترون است و به همین دلیل محصول PCR بزرگتری به شما می دهد. بهتر است گزینه “minimum size of the intron” را به ۲۰۰ جفت باز تغییر داد.
مطالب پیشنهادی: روش های مختلف استخراج RNA از سلول و بافت – RNA های غیر کد کننده
پارامترهای بررسی ویژگی جفت پرایمر ها
آخرین بخش در Primer-BLAST شامل تعداد زیادی پارامتر است که می توانید اکثر آنها را به حال خود رها کنید. مهمترین بخش این است که بررسی کنید کادر “Organism” حاوی نام یا شماره ارگانیسم صحیح باشد. در مثال ما، “۹۶۰۶” را در کادر داریم که کد Homo sapiens است.
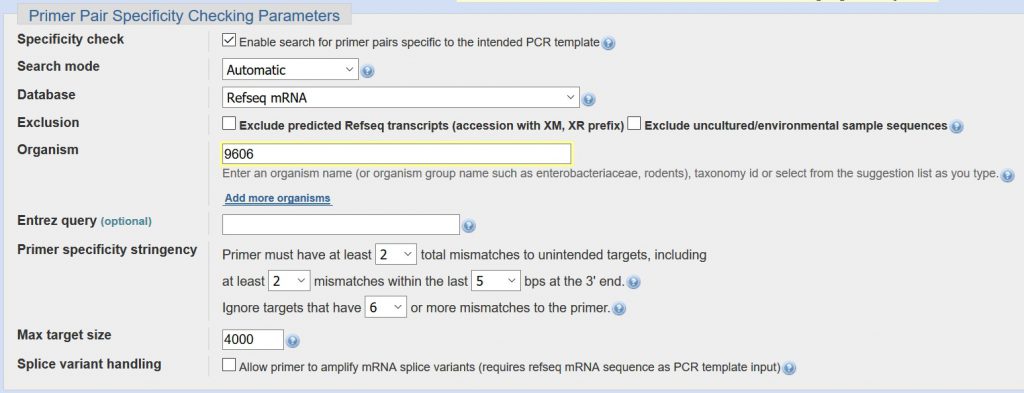
Primer-BLAST را اجرا کنید
پس از تنظیمات با کلیک بروی گزینه ی “Get Primers” نرم افزار Primer-BLAST اکنون از معیارهایی که شما مشخص کرده اید برای طراحی پرایمرهای PCR استفاده می کند. بسته به اینکه پارامترهای مورد نظر شما چقدر حساسیت داشته باشد، میتواند چند دقیقه طول بکشد.
خروجی پرایمر
پس از اجرای Primer-BLAST، پنجره خروجی نمایش داده می شود. در این حالت ما امیدواریم که برخی از پرایمرهای پیشنهاد شده کارایی کافی را برای pcr داشته باشند. خروجی را می توان به دو بخش تقسیم کرد: یک نمای گرافیکی از جفت پرایمر ها و یک گزارش با جزئیات از پرایمر ها. در مثال ما، بر اساس پارامترهایی که برای طراحی پرایمرهای mRNA IL-6 ارائه کردیم، ۱۰ جفت پرایمر داریم.
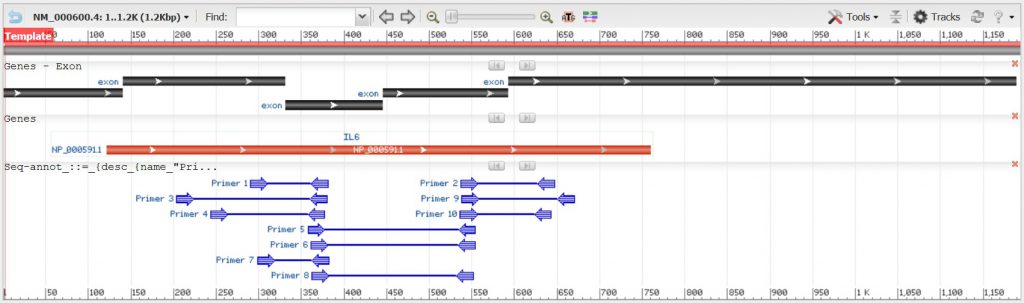
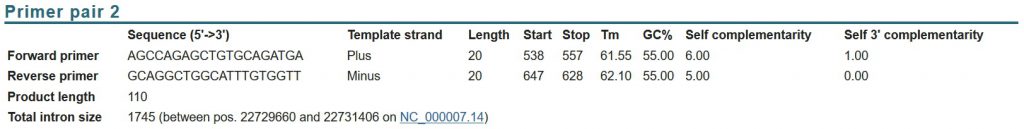
شناسایی بهترین پرایمرها
بطور خلاصه چند ویژگی مهم را در انتخاب توالی مورد نظر مطرح میکنیم:
اندازه محصول: ۷۰ تا ۲۰۰ جفت باز.
طول پرایمر: ۱۸–۲۲ نوکلئوتید (بهینه: ۲۰).
درصد محتوای GC: پرایمرها باید شامل ۵۰–۶۰ درصد از هر نوکلئوتید باشد.
پرایمر ها ترجیحا حاوی GC clamp باشد : گیره GC به وجود باز های G یا C در ۵ نوکلئوتید انتهای پرایمر گفته می شود. از آنجایی که بازهای G و C میل اتصال قوی تری دارند، این امر تضمین می کند که انتهای ‘۳ پرایمر به درستی با توالی متصل شود.
Tm: پرایمرها باید Tm نزدیک به همداشته باشند.
اجتناب از تکرار نوکلئوتیدی: از تکرار ۴ یا چند تکرار مجاور (مانند GTTTT) و یا تکرارهای دی نوکلئوتیدی (مانند GCGCGCGC) خودداری شود.
پرایمرها اگزون-اگزون باشد: پرایمر با یک اینترون بلند از هم جدا شود. این امر برای جلوگیری از تکثیر DNA ژنومی است.
انتخاب پرایمرهایی که نزدیک انتهای ‘۳ ژن متصل می شوند: این امر به ویژه در صورت استفاده از oligo(dT) در طول سنتز cDNA ، که منجر به اتصال به دم polyA در انتهای ‘۳ ژن میشود، مهم است. برخی از ژنهای بزرگ ممکن است هنگام استفاده از oligo(dT) به طور کامل تکثیر نشوند، بنابراین با داشتن پرایمرهایی که به انتهای ‘۳ متصل میشوند، کارایی پرایمر را می توان بهبود بخشید.
پایین self-complementarity score: پرایمرهایی را انتخاب کنید که امتیاز پایینی دارند، این امتیاز احتمال اتصال پرایمر به خود و به پرایمر دیگر را کم میکند.
پایین self 3’-complementarity score: پرایمرهایی را انتخاب کنید که دارای امتیاز کمتر از ۳ هستند، خود مکمل بودن ‘۳ این احتمال را می دهد که پرایمر به خود و به پرایمر دیگر در انتهای ‘۳ متصل شود. نمرات بالا نشان از افزایش این احتمال است
پرایمرهای اختصاصی ژن هدف شما
با توجه به نتایج برای جفت پرایمر ها، لیستی از “products on potentially unintended templates’ ” وجود دارد. این موارد نشان میدهد که ژن مورد نظر علاوه بر توالی مربوط به ژن ما ممکن است چه توالی های دیگری را تکثیر نماید. در اینجا به برخی از آنها اشاره می کنیم

- در تصویر بالا دو مورد اول در واقع دیگر رونوشتهای IL-6 هستند، که در مواردی که یک رونوشت خاص از ژن مورد نظر را لازم دارید، این مورد نباید وجود داشته باشد. در موارد عادی این مورد به عنوان یک ایراد محسوب نمیشود.
- در گزینه بعدی ژن دیگری (CYP1A1) وجود دارد که ممکن است با این پرایمرها نیز تکثیر شود. Primer-BLAST نشان میدهد که اندازه محصول در اینجا ۵۸ جفت باز است، که هنگام بردن محصول روی ژل آگارز باید به دنبال آن باشیم.
- در توالی بعدی احتمال اتصال پرایمر با توالی های مشابه دیگر را نشان میدهد که ممکن است در چند باز اختلاف داشته باشند. در مواردی ممکن است این توالی تکثیر شده و نتایج حاصل از PCR به صورت غیر اختصاصی ظاهر شود.
هیستوژنوتک امیدوار است با ارائه این مطلب به دانشجویان عزیز روند طراحی پرایمر qPCR با استفاده از NPrimer-BLAST را تسهیل نموده باشد. اگر تنظیمات بالا روی ژن مورد نظر شما کار نکرد، به عقب برگردید و برخی از تنظیمات را تغییر دهید. برای درک بهتر این مطالب لازم است به فرآیند ریل تایم پی سی آر آشنایی کامل داشته باشید.
مطالب مرتبط: اصول طراحی پرایمر – روش های مختلف استخراج RNA از سلول و بافت – سنتز cDNA از RNA استخراج شده از سلول و بافت
خدمات مرتبط: استخراج RNA و DNA سنتز cDNA ، طراحی پرایمر ، انجام تکنیک PCR و Real time PCR